Der EHEC-Erreger ist vermutlich durch das Verschmelzen zweier gefährlicher Keime entstanden
Kenne deinen Feind! So lautet eine Regel des chinesischen Militärstrategen Sun Tzu. Und sie gilt nicht nur bei Kriegen und Schlachten, sondern auch im Kampf gegen Infektionskrankheiten. Denn nur wenn Ärzte und Forscher über einen Krankheitserreger genau Bescheid wissen, können sie die Patienten optimal behandeln, die Quelle schnell eingrenzen, neue Infektionen verhindern.
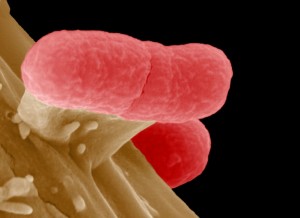 Deshalb war es für viele Ärzte und Forscher auch schon eine unangenehme Überraschung, als zu Beginn des EHEC-Ausbruchs klar wurde, dass es sich bei dem Übeltäter nicht um einen der üblichen Verdächtigen handelt, sondern um den seltenen Serotyp O104. „Dieser Erreger ist weltweit noch nie als Quelle eines Ausbruchs aufgefallen“, wunderte sich der EHEC-Forscher Helge Karch. Der Stamm war ihm in den vergangenen 15 Jahren nur einmal untergekommen. Das war 2001, bei einem schwer erkrankten Geschwisterpaar aus Köln.
Deshalb war es für viele Ärzte und Forscher auch schon eine unangenehme Überraschung, als zu Beginn des EHEC-Ausbruchs klar wurde, dass es sich bei dem Übeltäter nicht um einen der üblichen Verdächtigen handelt, sondern um den seltenen Serotyp O104. „Dieser Erreger ist weltweit noch nie als Quelle eines Ausbruchs aufgefallen“, wunderte sich der EHEC-Forscher Helge Karch. Der Stamm war ihm in den vergangenen 15 Jahren nur einmal untergekommen. Das war 2001, bei einem schwer erkrankten Geschwisterpaar aus Köln.
Inzwischen ist klar: Der Erreger ist noch viel ungewöhnlicher als gedacht. Denn während die Suche nach verunreinigten Lebensmitteln bisher keine klaren Erfolge gebracht hat, haben die Forscher einiges über ihren Gegner erfahren – auch durch die Hilfe hunderter Wissenschaftler, die die verfügbaren genetischen Daten nach Hinweisen durchforsten und ihre Ergebnisse über Blogs und Twitter verbreiten.
Den Anfang machte eine kleine Diskussion in der Kantine des Uniklinikums Hamburg-Eppendorf vor zwei Wochen. Eine Flut von Patientenproben schwappte damals auf die Forscher um den Mikrobiologen Holger Rohde zu. Beim Essen sprachen sie darüber, wie hilfreich es wäre, das Erbgut des Erregers untersuchen zu können. „Ich kenne da jemanden“, meldete sich ein chinesisch-stämmiger Däne, als Stipendiat an der Klinik, zu Wort. Er empfahl den Wissenschaftlern das Beijing Genomics Institute (BGI), eine riesige Sequenzierfabrik, die sich inzwischen im südchinesischen Shenzhen befindet. Hunderte der neuesten Sequenziermaschinen spucken dort täglich Billionen Buchstaben des Erbguts von Menschen, Tieren und Pflanzen aus. Die chinesischen Forscher haben das Genom der Gurke ebenso entziffert wie das Genom des Pandas. Wenige Orte wären in der Lage, das Erregergenom schneller zu sequenzieren. Das wusste auch Rohde.
„Wir haben dann etwas DNS des Erregers isoliert und am Mittwoch per Kurier nach Shenzhen geschickt“, erinnert sich Rohde. Freitag kam die Genomfracht in China an und am Wochenende begann die Arbeit. Nur wenige Tage später, meldeten die Chinesen: Das Erbgut ist entziffert – und machten es online für alle Welt verfügbar.
Tatsächlich handelte es sich dabei um tausende Bruchstücke DNS, die entziffert worden waren. Innerhalb von Stunden begann eine Armada von Forschern die Daten zusammenzufügen, die Sequenz mit denen anderer Erreger zu vergleichen und das ganze über Twitter und Blogs zu verbreiten. „Ich bin völlig überrascht, wie schnell das alles ging“, sagt Rohde. Alle paar Stunden schaue er online, was es Neues gibt.
Die Nachricht der Sequenzierung platzte auch in eine Konferenz über Erbgutsequenzierung und Epidemiologie, die gerade in England in der Nähe von Cambridge stattfand. Die anwesenden Wissenschaftler begannen ebenfalls sofort die vorhandenen Daten zu bearbeiten.
Die überraschendste Erkenntnis: Obwohl von Anfang an immer von einem EHEC-Keim gesprochen wurde, handelt es sich bei diesem Ausbruch genau genommen gar nicht um diesen Erreger. Genetisch gesehen müssen Escherichia coli-Bakterien zwei Gene in ihrem Erbgut haben, um als EHEC durchzugehen: Das Gen für das gefährliche Shigatoxin und das Gen für ein Eiweiß namens Intimin, das dem Bakterium hilft, sich im menschlichen Darm (vor allem bei Kindern) festzusetzen.
Intimin ist aber weder im Erbgut des jetzigen Keims noch in dem des Kölner Stammes vorhanden. Stattdessen weist das Erbgut der beiden Stämme zahlreiche Merkmale einer anderen Klasse von E.colis auf: EAEC, enteroaggregative E. coli.
Diese Erreger zeichnen sich unter anderem dadurch aus, dass sie Fimbrien bilden, winzige Tentakeln, mit denen sie sich im Darm besonders gut festsetzen können. „Diese Bakterien erzeugen üblicherweise Durchfälle, aber nicht die schweren blutigen Durchfälle und vor allem nicht das hämolytisch-urämische Syndrom“, sagt Martin Mielke vom RKI. Denn normalerweise können EAEC nicht das Shigatoxin herstellen. Das hat sich bei dem Stamm, der zurzeit sein Unwesen treibt, offenbar geändert.
„Die Sequenz sieht aus wie ein EAEC, der das Shigatoxin bekommen hat“, sagt Lothar Beutin vom Bundesinstitut für Risikobewertung. Die größte Ähnlichkeit haben die beiden deutschen Stämme mit einem EAEC-Erreger, der 2002 bei einem HIV-positiven Patienten in der zentralafrikanischen Republik gefunden wurde, der unter chronischem Durchfall litt. „Das könnte gewissermaßen der Großvaterstamm des jetzigen Erregers sein“, sagt Rohde.
Das wahrscheinlichste Szenario: Dieses oder ein sehr ähnliches Bakterium wurde von einem Phagen, einem Virus, das Bakterien befällt, infiziert. Das Virus schleuste dabei das Shigatoxin ein. „Wir können im Erbgut des Erregers noch Überreste dieses Phagen erkennen“, sagt Rohde.
Solche Gentransfers passieren in der Bakterienwelt ständig. Es ist also nur eine Frage der Zeit, bis eine besonders ungünstige Variante wie die jetzige herauskommt: Ein Bakterium, dass sich im Darm besonders gut festsetzen und vermehren kann und ein gefährliches Gift produziert.
Aber was bedeuten diese neuen Erkenntnisse? Zum einen finden sich EAEC-Stämme nicht im Magen-Darm-Trakt von Wiederkäuern, sondern beim Menschen, sagt Beutin. Damit scheidet Gülle als Ursprung der Kontamination höchstwahrscheinlich aus.
Da die Untersuchungen des RKI aber eindeutig auf Salate, Gurken und Tomaten hinweisen, stellt sich die Frage, wie der Erreger dann auf das Gemüse kam. Könnte es reichen, dass ein Mensch die Bakterien durch mangelnde Hygiene in die Lebensmittelkette einschleust? Ja, sagt Andreas Hensel, Präsident des Bundesinstituts für Risikobewertung. „Es gibt viele Beispiele, wo Menschen, die einen Krankheitserreger trugen aber nicht krank waren, die Keime in die Lebensmittelkette brachten.“ Ob das für den gegenwärtigen Erreger zutrifft, ist noch völlig unklar. Erst einmal gehe es darum, die unmittelbare Quelle des Ausbruchs zu finden, sagt Hensel.
Auch dabei könnten die neuen Erkenntnisse helfen. Denn EAEC-Keime können Biofilme bilden, mehrschichtige Bakterienteppiche, die regelrecht verklebt sind. „Dadurch können sie länger auf Oberflächen in der Umwelt überleben“, sagt Rohde. „Das ist zum Beispiel wichtig für die Experten, die jetzt in Bienenbüttel Proben sammeln und nach dem Erreger suchen“, sagt Hensel.
Trotzdem sprechen alle offiziellen Stellen weiterhin von einem EHEC-Bakterium. Das hat auch einen ganz pragmatischen Grund: Nur für EHEC-Keime gelten strikte Regularien wie etwa eine Meldepflicht. Würde das Bakterium nun umgetauft in EAEC, könnte das die Bekämpfung behindern, fürchten Seuchenschützer. Man kann seinen Feind offenbar auch zu gut kennen.
Leave A Comment